- Wie die Idee entstand, DNA für Rechenprobleme zu nutzen
- Der erste DNA-Computer
- Eine Zukunft mit vollständig organischen Rechnern?
- Anwendung des DNA-Computings in der Medizin
- Wie Forschende diese „lebenden Systeme“ einsetzen
- Erste Konstruktion eines genetischen Kippschalters
- Hilfe von der Evolution beim Computing
- Herausforderung: Mikroben programmieren
Rechnen mit DNA und programmierte Mikroben: Bio-Computing zeigt, was heute schon möglich ist

Seit Jahrhunderten grübeln Biologen und Philosophen darüber, was lebendige Wesen von toten Gegenständen wie Maschinen unterscheidet. Doch diese Unterscheidung könnte schon bald zunehmend irrelevant werden. Denn Biologie und Informationstechnologie wachsen im wahrsten Sinne des Wortes zusammen. In kaum einer Wissenschaft wird das so greifbar wie im Biological Computing, dem Rechnen mit biologischen Systemen.
Was mit dem Versuch begonnen hat, kurze DNA-Stücke zur Lösung schwieriger Rechenprobleme zu nutzen, hat sich zu einer wissenschaftlichen Disziplin entwickelt, die in die biochemischen Prozesse von Lebewesen eingreift, um sie zu programmieren wie Computer. Denn letztendlich sind auch Lebewesen für diese Forschenden einfach nur eine spezielle Art von Hardware. Jüngst zeigten Forschende von North Carolina State University und der Johns Hopkins University etwa, wie DNA zum Speichern von Daten und zum Lösen einfacher Aufgaben sowie für Sudoku- und Schachrätseln genutzt werden kann.
Dieser Text ist zuerst in der Ausgabe 4/2022 von MIT Technology Review erschienen. Darin beschäftigen wir uns mit den Möglichkeiten der Synthetischen Biologie. Hier könnt ihr die TR 4/2022 als Print- oder pdf-Ausgabe bestellen.
Wie die Idee entstand, DNA für Rechenprobleme zu nutzen
Es war ein Informatiker, der Anfang der 1990er-Jahre zuerst auf die Idee kam, DNA zu nutzen, um ein schwieriges Rechenproblem zu lösen – das sogenannte Hamiltonpfadproblem (HPP) in einem „gerichteten Graphen“. Leonard Adleman hatte Ende der 1970er-Jahre zusammen mit Ronald Rivest und Adi Shamir das RSA-Verschlüsselungssystem entwickelt, das heute weltweit eingesetzt wird. In den 80er-Jahren forschte er dann an Computerviren und deren Abwehr.
Beim HPP geht es darum, einen Pfad durch ein Netz aus Knoten zu finden, die miteinander verbunden sind. Und zwar so, dass jeder Knotenpunkt genau einmal besucht wird. In einem gerichteten Graphen dürfen als zusätzliche Schwierigkeit die Wege zwischen den Knoten nur in eine definierte Richtung begangen werden. Das bekannte Problem des Handlungsreisenden ist ein Spezialfall des Hamiltonproblems, bei dem nicht nur irgendeine Lösung gesucht wird, sondern der kürzestmögliche Pfad.
Adleman nutzte den Umstand aus, dass in der DNA jeweils nur spezifische Basenpaare aneinander binden – Adenin an Thymin und Cytosin an Guanin. Weil die molekularen Mechanismen beim Ablesen einer DNA ihn an das Verhalten abstrahierter Computermodelle – sogenannte Zustandsmaschinen – erinnerten, erschien es Adleman logisch, diese Moleküle zur Lösung schwieriger Informatikprobleme wie das HPP zu verwenden.
Der erste DNA-Computer
Unter Fachleuten sorgte Adlemans Verfahren für erhebliches Aufsehen, denn sein TT-100 genannter DNA-Computer, der aus 100 Mikrolitern DNA in einer Lösung bestand, konnte das Problem zwar nur für ein relativ überschaubares Netz mit sieben Knoten lösen. Für Informatiker ist das HPP aber interessant, weil es zu der Klasse von Problemen gehört, die als NP-komplett bezeichnet werden und damit zu den schwierigsten bekannten Problemen überhaupt. Der Aufwand, sie zu lösen, steigt exponentiell mit der Problemgröße. Mit seiner massiv-parallelen Informationsverarbeitung versprach der DNA-Computer nun, dass es möglich sein würde, solche NP-kompletten Probleme trotzdem zu knacken. Die Idee für den Algorithmus war zudem recht einfach:
- Erzeuge Einzelstränge aus DNA, die Knoten darstellen.
- Erzeuge zufällige Wege, die diese Knoten miteinander verbinden – hier kommt die komplementäre Bindung der Basen zum Tragen.
- Vermehre nur die Stränge, die den Start- und Endpunkt miteinander verbinden, um das Ergebnis leichter analysieren zu können.
- Sortiere alle Stränge aus, die zu lang (Knoten werden mehrfach besucht) oder zu kurz (nicht alle Knoten werden besucht) sind.
- Analysiere die Sequenz des Lösungspfades.
Einige Jahre später konnten Adleman und seine Kollegen auch die Lösung für ein erheblich komplexeres Problem zeigen: das sogenannte „Erfüllbarkeitsproblem“ (SAT) mit 20 Variablen. Dabei geht es um die Frage, ob es einen Satz logischer Variablen gibt, für die eine vorgegebene Reihe von logischen Verknüpfungen wahr, also erfüllt, ist. Doch trotz großer Hoffnungen auf molekularbiologische Fortschritte wie maßgeschneiderte Designer-Enzyme, mit denen die Reaktionen erheblich schneller ablaufen sollten, hat sich die Idee nicht durchgesetzt, denn sie hat zwei große Haken: Zum einen bestehen Software und Daten bei diesen Computern aus künstlich hergestellten DNA-Sequenzen, die für jedes Rechenproblem neu entworfen und hergestellt werden müssen.

Prinzip des DNA-Computers von Adleman: Jede Stadt wird durch eine spezifische Abfolge von 20 Basen repräsentiert. Ein Weg zwischen zwei Städten besteht aus zwei Teilstücken von je 10 Basen, die zu denen der ersten und denen der zweiten Stadt komplementär sind. (Bild: Manager Magazin)
Zum anderen hatten Adleman und Kollegen zwar die Hoffnung, dass man mit ihrer Methode auch Probleme mit wesentlich mehr Variablen lösen könnte – was nicht nur akademisch, sondern auch praktisch relevant gewesen wäre. Bereits 1995 veröffentlichte der Komplexitätsforscher Juris Hartmanis dazu jedoch eine recht ernüchternde Abschätzung: Er berechnete die kleinste DNA-Menge, die erforderlich ist, um alle möglichen Pfade durch einen Graphen zu kodieren, und kam zu dem Schluss, dass Adlemans Experiment bei einer Vergrößerung von sieben auf 200 Städte einen Anfangssatz von DNA-Strängen erfordern würde, der mehr wiegen würde als die Erde.
Eine Zukunft mit vollständig organischen Rechnern?
Diese Erkenntnis sollte sich in der Forschungscommunity immer stärker durchsetzen: Im Jahr 2000 schrieben die Informatiker Mitsunori Ogihara und der Molekularbiologe Animesh Ray, die beide selbst an DNA-Computern arbeiteten, in einem Kommentar für Nature: „Es ist töricht, zu versuchen, die Zukunft der Technologie vorherzusagen, aber es könnte sein, dass die ideale Anwendung für DNA-Berechnungen nicht in der Berechnung großer NP-Probleme liegt.“ Eines Tages könne es den Forschern zufolge jedoch einen Bedarf geben „für vollständig organische Rechengeräte, die in einem lebenden Körper die Signale aus verschiedenen Quellen integrieren und eine Reaktion in Form einer organischen Molekülabgabevorrichtung“ berechnen können.
„In den 90er-Jahren war die DNA-Berechnung sehr beliebt. Aber die meisten Leute in dieser Community waren besessen von mathematischen Problemen“, sagt auch Angel Goni-Moreno von der TU Madrid. „Aber das war nicht die richtige Anwendung für diese Technologie.“ Techniken des DNA-Computing würden jedoch neue Anwendungen ermöglichen, zum Beispiel in der Umweltsensorik oder der Medizin.
Anwendung des DNA-Computings in der Medizin
Pepijn Moerman und Rebecca Schulman von der Johns Hopkins University entwickelten 2020 zum Beispiel eine Methode, die mithilfe von DNA-Berechnungen anhand einer Blutprobe die Wahrscheinlichkeit angibt, ob der Patient an Krebs erkrankt ist. Die Forschenden identifizierten zunächst eine Reihe von microRNAs (miRNA) – kurze RNA-Moleküle, die als eine Art Fingerabdruck für die Erkennung von Lungenkrebs dienen könnten. Anschließend nutzten sie klinische Daten über die Konzentrationen dieser miRNAs bei gesunden Personen und Lungenkrebspatienten. Mit diesen Daten trainierten sie Software darauf, zu erkennen, welche miRNA für das Auftreten von Lungenkrebs besonders aussagekräftig ist. Die Software identifizierte vier besonders deutliche miRNA-Marker: zwei, die eine erhöhte Krebswahrscheinlichkeit signalisieren (Bad-news), und zwei, bei denen eine hohe Konzentration darauf hindeutet, dass ein Patient gesund ist (Good-news).
Ein kleiner DNA-Rechner berechnet nun aus Proben von Patienten, mit welcher Wahrscheinlichkeit sie an Lungenkrebs erkranken. Der „molekulare Klassifikator“ besteht aus zwei aufeinanderfolgenden Berechnungen: Zunächst wird jedes Eingangssignal – also jeder miRNA-Marker – mit einem Gewichtungsfaktor multipliziert, indem die miRNA-Moleküle mit einem DNA-Komplex interagieren, der für jeden eingehenden RNA-Strang eine gewichtete Anzahl DNA-Output-Stränge freisetzt. Paare von Good-news- und Bad-news-DNA-Strängen koppeln zusammen. Nur derjenige Strang, der im Überschuss vorhanden ist, reagiert mit einem Fluoreszenzmarker und löst ein entsprechendes Signal aus.
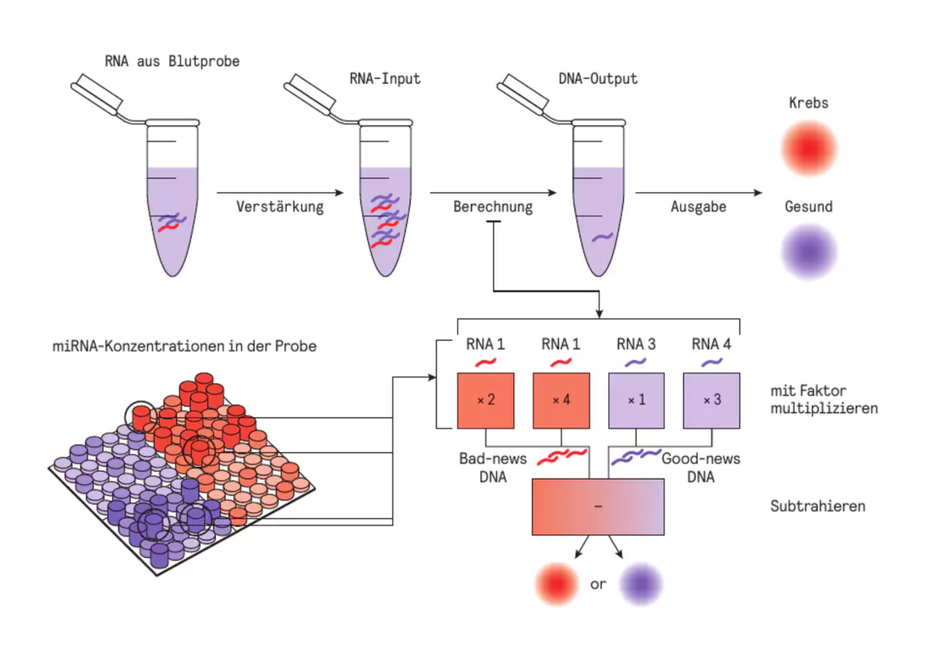
Forschende der Johns Hopkins University entwickelten eine Methode, die mithilfe von DNA-Berechnungen anhand einer Blutprobe die Wahrscheinlichkeit angibt, ob der Patient an Krebs erkrankt ist. Die Idee: Mikro-RNAs, die als Marker für Krebs oder Gesundheit dienen, lösen die Freisetzung von entsprechenden DNA-Strängen aus. Stränge, die Krankheit und solche, die Gesundheit signalisieren, lagern sich aneinander. Die jeweils überzähligen Stränge lösen ein Leuchtsignal aus. (Bild: Nature Nanotechnology / Pepijn G. Moerman / Rebecca Schulman)
Im Grunde ist das, was der DNA-Rechner von Moerman und Schulman da macht, die Berechnungen eines einfachen neuronalen „Netzes“ mit einem Neuron nachzubilden. Denn auch in jedem Neuron eines künstlichen neuronalen Netzes werden die Input-Größen mit einem Gewichtungsfaktor multipliziert und anschließend addiert. Auf einem ganz ähnlichen Prinzip beruht ein Biosensor, der Schadstoffe im Wasser anzeigt. Das Gerät, das von Forschenden der Northwestern University entwickelt wurde, enthält sieben Teströhrchen, in denen DNA so miteinander verknüpft wird, dass bei einer bestimmten Schadstoffkonzentration ein fluoreszierendes Protein hergestellt wird. Je mehr Röhrchen leuchten, desto größer die Konzentration der Schadstoffe.
Wie Forschende diese „lebenden Systeme“ einsetzen
Für Goni-Moreno sind solche Berechnungen mit DNA-Molekülen aber „nur ein Anfang, keinesfalls das Ziel“, sagt er. „Lebende Systeme können Input-Signale verarbeiten, mit denen ein herkömmlicher Computer nichts anfangen kann: Chemikalien, Krankheitserreger, Stressfaktoren – all diese Arten von Informationen sind brandneu. Aber auch die Art und Weise, wie diese Informationen verarbeitet werden, ist vollkommen anders als in herkömmlichen Rechnern, denn lebende Systeme können sich anpassen, sich entwickeln – es gibt eine natürliche Arbeitsteilung bei ihnen“, schwärmt Goni-Moreno.
Tatsächlich verwenden immer mehr Forschungsgruppen mittlerweile diesen – eher indirekten – Weg des biologischen Rechnens: Sie nutzen die biologischen Mechanismen innerhalb lebender Zellen, um chemische Input-Signale logisch miteinander zu verknüpfen, sodass entsprechende Output-Substanzen erzeugt werden. Diese können wiederum als neue Input-Signale an anderer Stelle verwendet werden.
Die Idee gibt es schon seit längerer Zeit: Bereits 1970 schrieben die Molekularbiologen Francois Jacob und Jaques Monod, dass biologische Systeme „nach den Prinzipien der Algebra von George Boole“ funktionierten, „ähnlich wie Computer“ – eine Folgerung aus ihrer früheren Entdeckung über die Funktion des Laktose-Abbaus durch das Bakterium E. coli. Denn E. coli. produziert das entsprechende Enzym zum Abbau der Laktose nur, wenn diese vorhanden ist. Wenn das Bakterium aber auf einem Nährboden wächst, der auch Glukose enthält, verarbeitet es zunächst die energiereichere Glukose. Der DNA-Abschnitt, der diese Regulation kodiert, wird Lac-Operon genannt.
Die Regulation der Gene, die für die Produktion der entsprechenden Enzyme verantwortlich sind, könnten aber auch als das Verhalten einer einfachen Maschine gedacht werden. Zumindest, wenn man an dieser Stelle wie ein Informatiker denkt. Für die lässt sich das, was E.coli tut, mit dem Modell des Zustandsautomaten beschreiben: Das ist ein fiktiver Automat, der verschiedene Eingaben akzeptiert, und abhängig von diesen Eingaben in einen neuen Zustand übergeht. Das macht er so lange, bis er seinen Endzustand erreicht hat.
Solche Zustandsautomaten haben die praktische Eigenschaft, dass sich aus der Auflistung aller möglichen Zustände, Inputs und Übergänge mit ein paar einfachen mathematischen Tricks ein Schaltwerk ableiten lässt, das aus logischen Gattern und Zustandsspeichern besteht und das gewünschte Verhalten zeigt. Die Anweisungen, die das Verhalten dieser Maschinen steuern, sind in diesem Schaltwerk hart verdrahtet. In herkömmlichen Computern bestehen diese Gatter und Zustandsspeicher aus Transistoren – das ist aber längst nicht die einzige Möglichkeit.
Erste Konstruktion eines genetischen Kippschalters
Es sollte jedoch noch rund 30 Jahre dauern, bis die Forschung in der Lage war, so etwas wie einen Zustandsautomaten auch in biologischen Systemen zu nutzen. 2000 erschienen zwei bahnbrechende wissenschaftliche Arbeiten dazu. Die erste war eine Arbeit von Timothy Gardner, Charles Cantor und Jim Collins von der Boston University: Gardner und Kollegen konstruierten eine Art genetischen Kippschalter. Das Team wählte zwei Gene, die sich gegenseitig hemmen – das heißt, jedes produziert ein Molekül, das das jeweils andere Gen ausschaltet, wenn es aktiviert wird. Zur Aktivierung der beiden Gene musste jeweils ein spezieller Stimulus vorhanden sein – bei dem einen Gen eine Chemikalie, bei dem anderen eine Temperaturänderung.

Forschende der Northwestern University entwickelten einen Biosensor, der mithilfe von DNA-Logik direkt die Konzentration von Schadstoffen in Wasser anzeigt. Je mehr Röhrchen leuchten, desto belasteter ist das Wasser. (Bild: Northwestern University)
Bemerkenswert daran war, dass diese Stimuli nicht kontinuierlich angewendet werden mussten – ein kurzer scharfer Impuls reichte aus, um das System in einen von zwei Zuständen zu kippen. Die Verknüpfung der Gene verhielt sich wie eine Flip-Flop-Schaltung – ein bistabiles System, das aus einem Zustand in einen anderen Zustand kippt und dort so lange bleibt, bis der nächste Befehl zum Umschalten kommt. Wie bei den anderen Experimenten verwendeten Gardner und seine Kollegen ein grün fluoreszierendes Protein als „Anzeige“ für den Systemzustand. In einem Zustand leuchteten die Zellen, im anderen nicht.
Im selben Jahr konstruierten Michael B. Elowitz und Stanislas Leibler von der Princeton University ein synthetisches Netzwerk zur Genregulation, das wie ein Schwingkreis funktionierte – sie nannten diese Konstruktion einen Repressilator. Dafür nutzten sie drei Gene in Bakterien, von denen jedes eines der anderen Gene unterdrückte oder ausschaltete: Das Produkt von Gen A schaltet das Gen B aus. Das Fehlen des normalerweise von Gen B codierten Proteins, das eigentlich Gen C unterdrückt, bewirkt, dass Gen C aktiviert wird. Das Produkt von Gen C schaltet Gen A wieder aus – und wo weiter. Um zu testen, ob ihre Idee funktioniert, ließen die Forschenden eine Bakterienpopulation grün leuchtend blinken wie aufdringlichen Weihnachtsschmuck. Wie in elektronischen Schaltungen ließe sich solch ein Oszillator auch in biologischen Schaltungen als Taktgeber einsetzen.
Hilfe von der Evolution beim Computing
Goni-Moreno ist sich ziemlich sicher, dass diese spezielle Art des Computing der von klassischen Computern überlegen sein kann – allerdings nur für bestimmte Anwendungen. Ähnlich wie ein Quantencomputer niemals einen herkömmlichen Rechner ersetzen wird, bestimmte Probleme aber knackt, an denen sich klassische Computer die Zähne ausbeißen, gäbe es für biologische Computer Anwendungen, in denen sie ihre „Cellular Supremacy“, wie er das nennt, ausspielen können. Danach gefragt, welche das sein könnten, bleibt Goni-Moreno, der zunächst Informatik studierte, bevor er zur Synthetischen Biologie wechselte, recht vage: „Wenn ein Ökosystem beschädigt ist oder droht zu kollabieren, dann wollen wir Mikroben so programmieren, dass sie erkennen, wo das Problem liegt, und das System wieder ins Gleichgewicht bringen“, sagt Goni-Moreno. Er und sein Team arbeiten mit Pseudomonis Putida, stäbchenförmigen Bakterien, die im Boden leben und in der Lage sind, mit Naphtalen (ein polyzyklischer aromatischer Kohlenwasserstoff) verseuchte Böden zu reinigen. 2020 hatte ein Forschungsteam aus Singapur gezeigt, dass man Bakterien dazu bringen kann, Quecksilber anzureichern – allerdings hatten die Mikroben sich dabei selbst vergiftet. Goni-Moreno und Kollegen wollen die Fähigkeiten ihrer Bakterien verbessern, „die natürlichen Stoffwechselprozesse feintunen“ und auf andere Schadstoffe ausweiten, aber so, dass sie am Leben bleiben.
Herausforderung: Mikroben programmieren
Ein weiteres Problem, an dem Goni-Moreno und sein Team arbeiten, ist die sogenannte Stickstofffixierung. „Wir wollen Mikroben programmieren, um Pflanzen bei der Stickstofffixierung zu helfen. Das gibt es zwar auch schon in der Natur, aber unser Ziel ist es, Umfang und Geschwindigkeit zu erhöhen.“ Zwar gäbe es bereits jetzt Unternehmen wie Pivot Bio, die solche Organismen entwickelt hätten. „Aber das ist nur eine Spezies“, sagt er. „Natürliche Mikrobiome sind erstaunlich robust, weil sie Hunderte von Arten enthalten. Wir müssen uns dem annähern, was die Natur tut.“
Denn auch in der Natur sei der Umfang dessen, was ein einzelner Organismus leisten könnte, begrenzt. „Wenn das Programm, das man in die Zelle einbaut, zu groß wird, kann man die Zelle beschädigen. Deshalb ist es gut, es in verschiedene Teile zu packen.“ Zudem böte eine komplette Population programmierter Bakterien die Möglichkeit, einen Mechanismus zu nutzen, der für die Synthetische Biologie Fluch und Segen zugleich ist: die Evolution. „Die Evolution ist die Antwort der Natur auf die Tatsache, dass biologische Prozesse mit Fehlern behaftet sind, mit Rauschen“, sagt er. „Rauschen ermöglicht lebenden Systemen, sich weiterzuentwickeln.“ Das bedeute aber auch, dass die Herausforderung darin bestünde, die Organismen so zu programmieren, dass eine evolutionäre Entwicklung die Programmierung nicht zerstört. Wie das gehen kann? „Ich habe ein paar Ideen“, sagt Goni-Moreno, will aber keine Einzelheiten verraten. Nur so viel: „Ich denke, die Biologie kann hier viel von der Informatik lernen.“






